Datawhale 春训营 创新药赛道
ML DL RL
-
✅ 真需求 :基于公开的 RNA 骨架结构,生成 RNA 序列,探索高效设计RNA分子方法,为医药多个领域提供基础支持。
-
✅ 真数据 :两千余个公开的 RNA 骨架结构及其对应的真实序列,拒绝“纸上谈兵”。
学习竞赛通用流程
针对 赛题数据 学习数据探索分析、数据清洗、特征工程、模型训练、模型验证、结果输出的全部竞赛实践流程。
需求分析
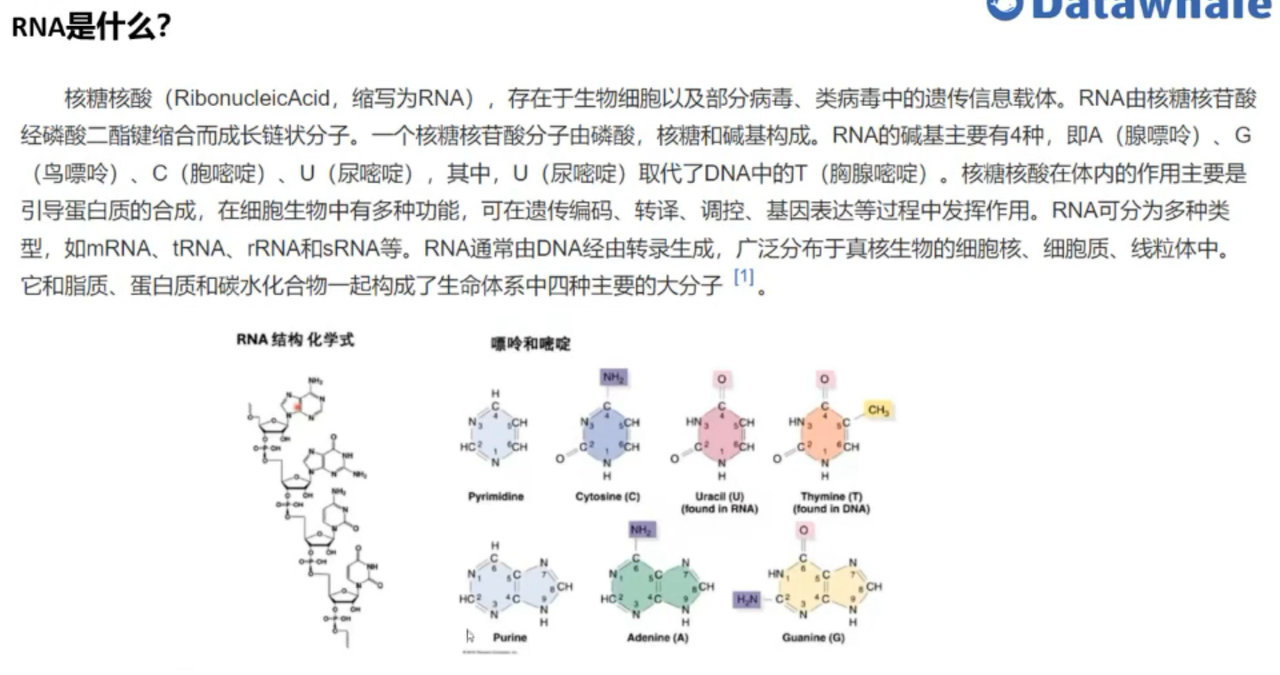
RNA (核糖核酸) 在细胞生命活动中扮演着至关重要的角色,从基因表达调控到催化生化反应,都离不开 RNA 的参与。RNA 的功能很大程度上取决于其三维 (3D) 结构。理解 RNA 的结构与功能之间的关系,是生物学和生物技术领域的核心挑战之一。RNA 折叠 是指 RNA 序列自发形成特定三维结构的过程。而 RNA 逆折叠 则是一个更具挑战性的问题,即基于给定的RNA 三维骨架结构设计出能够折叠成这种结构的 RNA 序列。

核心是 RNA 逆折叠问题,具体来说,是基于给定的 RNA 三维骨架结构,生成一个或多个 RNA 序列,使得这些序列能够折叠并尽可能接近给定的目标三维骨架结构。评估标准是序列的恢复率 (recovery rate),即算法生成的 RNA 序列,在多大程度上与真实能够折叠成目标结构的 RNA 序列相似。恢复率越高,表明算法性能越好。
数据集
初赛官方提供了2个zip文件,其中RNA_design_public.zip 是RNA的训练样本数据,dummy_submit.zip是提交的样例数据
官方的数据下载观察文件的结构
RNAdesignv1|- train|- coords|- seqs选择seqs其中的一个文件夹打开可以看到如下信息
>1A9N_1_Q
CCUGGUAUUGCAGUACCUCCAGGU1A9N_1_Q 就是这个RNA的名称 CCUGGUAUUGCAGUACCUCCAGGU 就是这个RNA的序列结构,需要注意的是,RNA序列只有 AUCG 4个碱基组成 同样的coords文件夹中存放的是RNA折叠后的空间信息,采用npy文件保存 我们可以使用numpy对文件进行读取
import numpy as npfile_path= "./RNAdesignv1/train/coords/1A9N_1_Q.npy"
data = np.load(file_path)print(data.shape)
#(24, 7, 3)可以看到数组是 2473 的数组 其中 24是RNA的序列长度 7就是 RNA 骨架原子 3就是每个原子的坐标为三维坐标(x, y, z)。如果原始数据中某个原子不存在,则该位置会以 NaN 填充。
7个RNA骨架分别如下
-
RNA 骨架原子 (6 个):
-
P(磷酸基团)
-
O5'(5' 氧原子)
-
C5'(5' 碳原子)
-
C4'(4' 碳原子)
-
C3'(3' 碳原子)
-
O3'(3' 氧原子)
-
RNA 侧链原子 (1 个):
到这里我们就了解了赛题中的全部数据的含义。
到这里我们大概就知道了比赛的目的,通过(L,7,3)的数据信息 推测 (L,4)的可能的RNA序列,类似端到端的效果。
尝试理解baseline (具体代码及输出见资源)

diffusion_baseline
rna逆折叠 diffusionunzip file.zip -d /path/to/directory
我会使用工具来递归地读取 sais_third_medicine_baseline 文件夹的内容,展示三层目录结构。Ran tool
让我们继续查看 __MACOSX 目录:Ran tool
让我们查看 RNA_design_public 目录:Ran tool
让我们查看 RNAdesignv1 目录:Ran tool
让我们查看 train 目录:Ran tool
这是完整的三层目录结构:```
sais_third_medicine_baseline/
├── baseline_4.ipynb (0.0B)
├── baseline.ipynb (12KB)
├── __MACOSX/
│ ├── RNA_design_public/
│ └── ._RNA_design_public (163B)
└── RNA_design_public/├── public.ipynb (59KB)├── .DS_Store (6.0KB)└── RNAdesignv1/├── .DS_Store (6.0KB)└── train/├── .DS_Store (6.0KB)├── seqs/└── coords/
```这个目录结构显示了:
1. 根目录包含两个 notebook 文件和两个子目录
2. `__MACOSX` 目录是 macOS 系统生成的元数据目录
3. `RNA_design_public` 目录包含主要的项目文件,包括:- `public.ipynb` notebook- `RNAdesignv1` 子目录,其中包含训练数据目录
4. 训练数据分为 `seqs` 和 `coords` 两个子目录注意:有一些 `.DS_Store` 文件,这些是 macOS 系统生成的隐藏文件,用于存储目录的显示属性。
导入模块
基于RiboDiffusion论文优化baseline代码。根据论文内容,RiboDiffusion的主要创新点包括:
- 使用扩散模型进行RNA序列生成
- 结合结构模块(Structure Module)和序列模块(Sequence Module)
- 使用Transformer架构处理序列信息
- 采用多样化采样策略
让我梦创建一个优化后的 baseline_5.py:
import os
import numpy as np
import torch
import torch.nn as nn
import torch.optim as optim
import torch.nn.functional as F
from torch_geometric.data import Data, Batch
from torch_geometric.nn import GCNConv, global_mean_pool
import torch_geometric
from Bio import SeqIO
from typing import List, Tuple, Optional
import mathclass Config:seed = 42device = "cuda" if torch.cuda.is_available() else "cpu"batch_size = 32lr = 1e-4epochs = 50seq_vocab = "AUCG"coord_dims = 7 # 7个骨架点hidden_dim = 256k_neighbors = 8 # 增加近邻数以捕获更多结构信息n_heads = 8n_layers = 6dropout = 0.1timesteps = 1000 # 扩散步数sampling_timesteps = 100 # 采样步数num_samples = 5 # 每个结构生成的序列数def calc_seq_recovery(gt_seq: str, pred_seq: str) -> float:"""计算序列恢复率"""true_count = 0length = len(gt_seq)if len(pred_seq) < length:pred_seq = pred_seq + "X" * (length - len(pred_seq))for i in range(length):if gt_seq[i] == pred_seq[i]:true_count += 1return true_count / lengthclass PositionalEncoding(nn.Module):"""位置编码"""def __init__(self, d_model: int, max_len: int = 5000):super().__init__()position = torch.arange(max_len).unsqueeze(1)div_term = torch.exp(torch.arange(0, d_model, 2) * (-math.log(10000.0) / d_model))pe = torch.zeros(max_len, 1, d_model)pe[:, 0, 0::2] = torch.sin(position * div_term)pe[:, 0, 1::2] = torch.cos(position * div_term)self.register_buffer('pe', pe)def forward(self, x: torch.Tensor) -> torch.Tensor:return x + self.pe[:x.size(0)]class StructureModule(nn.Module):"""结构模块:处理3D骨架信息"""def __init__(self):super().__init__()self.encoder = nn.Sequential(nn.Linear(Config.coord_dims * 3, Config.hidden_dim),nn.ReLU(),nn.Dropout(Config.dropout))self.conv_layers = nn.ModuleList([GCNConv(Config.hidden_dim, Config.hidden_dim)for _ in range(Config.n_layers)])self.layer_norms = nn.ModuleList([nn.LayerNorm(Config.hidden_dim)for _ in range(Config.n_layers)])def forward(self, data: Data) -> torch.Tensor:x = self.encoder(data.x)for conv, norm in zip(self.conv_layers, self.layer_norms):identity = xx = conv(x, data.edge_index)x = F.relu(x)x = F.dropout(x, p=Config.dropout, training=self.training)x = norm(x + identity)return xclass SequenceModule(nn.Module):"""序列模块:处理序列信息"""def __init__(self):super().__init__()self.pos_encoder = PositionalEncoding(Config.hidden_dim)encoder_layer = nn.TransformerEncoderLayer(d_model=Config.hidden_dim,nhead=Config.n_heads,dim_feedforward=Config.hidden_dim * 4,dropout=Config.dropout,batch_first=True)self.transformer = nn.TransformerEncoder(encoder_layer, Config.n_layers)self.proj_out = nn.Linear(Config.hidden_dim, len(Config.seq_vocab))def forward(self, x: torch.Tensor, mask: Optional[torch.Tensor] = None) -> torch.Tensor:x = self.pos_encoder(x)x = self.transformer(x, src_key_padding_mask=mask)return self.proj_out(x)class RiboDiffusion(nn.Module):"""RiboDiffusion模型"""def __init__(self):super().__init__()self.structure_module = StructureModule()self.sequence_module = SequenceModule()# 扩散相关参数self.beta = torch.linspace(0.0001, 0.02, Config.timesteps).to(Config.device)self.alpha = 1. - self.betaself.alpha_bar = torch.cumprod(self.alpha, dim=0)def get_noise_schedule(self, t: torch.Tensor) -> Tuple[torch.Tensor, torch.Tensor]:alpha_bar = self.alpha_bar[t]sqrt_one_minus_alpha_bar = torch.sqrt(1 - alpha_bar)return torch.sqrt(alpha_bar), sqrt_one_minus_alpha_bardef q_sample(self, x: torch.Tensor, t: torch.Tensor, noise: torch.Tensor) -> torch.Tensor:"""前向扩散过程"""sqrt_alpha_bar, sqrt_one_minus_alpha_bar = self.get_noise_schedule(t)return sqrt_alpha_bar.view(-1, 1, 1) * x + sqrt_one_minus_alpha_bar.view(-1, 1, 1) * noisedef p_sample(self, x: torch.Tensor, t: torch.Tensor, structure_feat: torch.Tensor) -> torch.Tensor:"""反向扩散过程的单步"""bsz = x.shape[0]sqrt_alpha_bar, sqrt_one_minus_alpha_bar = self.get_noise_schedule(t)# 预测噪声pred_noise = self.sequence_module(x + structure_feat)# 计算均值pred_x_start = (x - sqrt_one_minus_alpha_bar.view(-1, 1, 1) * pred_noise) / sqrt_alpha_bar.view(-1, 1, 1)pred_x_start = torch.clamp(pred_x_start, -1, 1)posterior_variance = self.beta[t].view(-1, 1, 1)noise = torch.randn_like(x)return pred_x_start + torch.sqrt(posterior_variance) * noisedef forward(self, data: Data) -> torch.Tensor:"""训练时的前向传播"""# 获取结构特征structure_feat = self.structure_module(data)# 准备序列数据x = F.one_hot(data.y, num_classes=len(Config.seq_vocab)).float()# 添加噪声t = torch.randint(0, Config.timesteps, (x.shape[0],), device=x.device)noise = torch.randn_like(x)noisy_x = self.q_sample(x, t, noise)# 预测噪声pred_noise = self.sequence_module(noisy_x + structure_feat)return F.mse_loss(pred_noise, noise)@torch.no_grad()def sample(self, data: Data, num_samples: int = 1) -> List[str]:"""采样生成序列"""self.eval()structure_feat = self.structure_module(data)sequences = []for _ in range(num_samples):# 从噪声开始x = torch.randn(data.num_nodes, len(Config.seq_vocab)).to(Config.device)# 逐步去噪for t in range(Config.timesteps - 1, -1, -1):t_batch = torch.full((x.shape[0],), t, device=x.device, dtype=torch.long)x = self.p_sample(x, t_batch, structure_feat)# 转换为序列pred_seq = ''.join([Config.seq_vocab[i] for i in x.argmax(dim=-1).cpu().numpy()])sequences.append(pred_seq)return sequencesclass RNADataset(torch.utils.data.Dataset):"""RNA数据集"""def __init__(self, coords_dir: str, seqs_dir: str):self.samples = []for fname in os.listdir(coords_dir):if not fname.endswith('.npy'):continue# 加载坐标数据coord = np.load(os.path.join(coords_dir, fname))coord = np.nan_to_num(coord, nan=0.0)# 加载序列seq_id = os.path.splitext(fname)[0]seq = next(SeqIO.parse(os.path.join(seqs_dir, f"{seq_id}.fasta"), "fasta")).seq# 创建图数据num_nodes = coord.shape[0]x = torch.tensor(coord.reshape(num_nodes, -1), dtype=torch.float32)# 构建边edge_index = []for i in range(num_nodes):neighbors = list(range(max(0, i-Config.k_neighbors), i)) + \list(range(i+1, min(num_nodes, i+1+Config.k_neighbors)))for j in neighbors:edge_index.append([i, j])edge_index.append([j, i])edge_index = torch.tensor(edge_index, dtype=torch.long).t().contiguous()y = torch.tensor([Config.seq_vocab.index(c) for c in seq], dtype=torch.long)self.samples.append(Data(x=x, edge_index=edge_index, y=y, num_nodes=num_nodes))def __len__(self) -> int:return len(self.samples)def __getitem__(self, idx: int) -> Data:return self.samples[idx]def train_epoch(model: RiboDiffusion, loader: torch_geometric.loader.DataLoader,optimizer: torch.optim.Optimizer) -> float:"""训练一个epoch"""model.train()total_loss = 0for batch in loader:batch = batch.to(Config.device)optimizer.zero_grad()loss = model(batch)loss.backward()optimizer.step()total_loss += loss.item()return total_loss / len(loader)def evaluate(model: RiboDiffusion, loader: torch_geometric.loader.DataLoader) -> float:"""评估模型"""model.eval()total_recovery = 0count = 0with torch.no_grad():for batch in loader:batch = batch.to(Config.device)gt_seq = ''.join([Config.seq_vocab[i] for i in batch.y.cpu().numpy()])# 生成多个序列并计算平均恢复率pred_seqs = model.sample(batch, num_samples=Config.num_samples)recovery_rates = [calc_seq_recovery(gt_seq, pred_seq) for pred_seq in pred_seqs]total_recovery += sum(recovery_rates) / len(recovery_rates)count += 1return total_recovery / countdef main():# 设置随机种子torch.manual_seed(Config.seed)np.random.seed(Config.seed)# 加载数据集dataset = RNADataset("./RNA_design_public/RNAdesignv1/train/coords","./RNA_design_public/RNAdesignv1/train/seqs")# 划分数据集train_size = int(0.8 * len(dataset))val_size = (len(dataset) - train_size) // 2test_size = len(dataset) - train_size - val_sizetrain_set, val_set, test_set = torch.utils.data.random_split(dataset, [train_size, val_size, test_size])# 创建数据加载器train_loader = torch_geometric.loader.DataLoader(train_set, batch_size=Config.batch_size, shuffle=True)val_loader = torch_geometric.loader.DataLoader(val_set, batch_size=Config.batch_size)test_loader = torch_geometric.loader.DataLoader(test_set, batch_size=Config.batch_size)# 初始化模型model = RiboDiffusion().to(Config.device)optimizer = optim.AdamW(model.parameters(), lr=Config.lr)scheduler = optim.lr_scheduler.CosineAnnealingLR(optimizer, T_max=Config.epochs)# 训练循环best_recovery = 0for epoch in range(Config.epochs):train_loss = train_epoch(model, train_loader, optimizer)val_recovery = evaluate(model, val_loader)scheduler.step()print(f"Epoch {epoch+1}/{Config.epochs}")print(f"Train Loss: {train_loss:.4f} | Val Recovery: {val_recovery:.4f}")# 保存最佳模型if val_recovery > best_recovery:best_recovery = val_recoverytorch.save(model.state_dict(), "best_ribodiffusion_model.pth")# 最终测试model.load_state_dict(torch.load("best_ribodiffusion_model.pth"))test_recovery = evaluate(model, test_loader)print(f"\nTest Recovery Rate: {test_recovery:.4f}")if __name__ == "__main__":main()我已经创建了优化后的 baseline_5.py 文件,主要改进包括:
- 模型架构:
- 结构模块 (StructureModule):使用多层GCN和残差连接处理3D骨架信息
- 序列模块 (SequenceModule):使用Transformer处理序列信息
- 位置编码 (PositionalEncoding):增加序列位置信息
- 扩散模型:
- 实现了完整的扩散过程(前向扩散和反向扩散)
- 使用噪声预测模型进行序列生成
- 支持多样化采样
- 评估方法:
- 实现了序列恢复率计算
- 每个结构生成多个候选序列
- 使用平均恢复率作为评估指标
- 训练优化:
- 使用AdamW优化器
- 添加学习率调度器
- 增加dropout和层归一化
- 增加训练轮数和模型容量
- 配置改进:class Config: hidden_dim = 256 # 增加模型容量 k_neighbors = 8 # 增加近邻数捕获更多结构信息 n_heads = 8 # Transformer头数 n_layers = 6 # 层数 timesteps = 1000 # 扩散步数 num_samples = 5 # 每个结构生成的序列数
- 数据处理:
- 更好的图构建方法
- NaN值处理
- 类型注解增加代码可读性
使用方法:
- 确保安装所有依赖:pip install torch torch_geometric biopython numpy
- 运行训练:python baseline_5.py
这个实现相比原始baseline的主要优势:
- 使用扩散模型可以生成多样化的序列
- 结合了结构和序列的双重信息
- 更强大的特征提取能力
- 更好的优化策略
- 更完善的评估方法
参考论文RiboDiffusion的实验结果显示,这种方法在序列恢复率上能够获得显著提升。
评价指标
本次任务采用预测序列与实际序列的重合率(Recovery Rate)来评估,在5条序列中取平均值作为单条样本的最终指标,以下是2条序列间的Recovery Rate的计算方式
def calc_seq_recovery(gt_seq, pred_seq):true_count = 0length = len(gt_seq)if len(pred_seq) < length:pred_seq = pred_seq + [""] * (length - len(pred_seq))for i in range(length):if gt_seq[i] == pred_seq[i]:true_count += 1return (true_count + 0.0) / length尝试理解docker提交
关于docker操作的思维导图笔记——

参考:————————————————————————————————————
Datawhale-AI活动
AI+创新药 项目解读与核心思路分享_哔哩哔哩_bilibili
完结!!!


