AI 驱动抗生素发现:从靶点到化合物测试
AI 驱动抗生素发现:从靶点到化合物测试
目录
- 基于 AI 驱动的研发流程发现抗生素,整合靶点选择和深度学习分子生成,显著提升了候选药物发现效率。
- 结合数据平衡技术,机器学习和 AutoML 能有效提升不平衡数据集分类性能。
- RibbonFold 是一种新的 AI 模型,可以准确预测淀粉样蛋白的多态性纤维结构,为阿尔茨海默症等疾病研究提供新工具。
- duAb 利用语言模型设计的肽段引导去泛素酶,实现对“不可成药”蛋白的精准稳定,为蛋白质组编辑提供新策略。
- RetroGFN 利用 GFlowNet 实现更具多样性和可行性的逆合成反应预测,提升药物研发效率。
1. AI 驱动抗生素发现:从靶点到化合物
本研究提出了一种端到端的 AI 驱动抗生素发现流程,整合了基于结构的靶点选择和深度学习驱动的分子生成、筛选和化合物实现。研究者首先利用 Foldseek 对七种病原体的预测蛋白质组进行大规模结构聚类,以识别保守的、必需的、非人类同源的蛋白质,这些蛋白质通常会被基于序列的方法遗漏。选择的靶点包括已验证的抗生素靶点 MurC、新型酶靶点 CdsA 和推定的二聚化界面靶点 CohE,它们与已知数据集的结构相似性各不相同,以便严格评估模型的泛化能力。
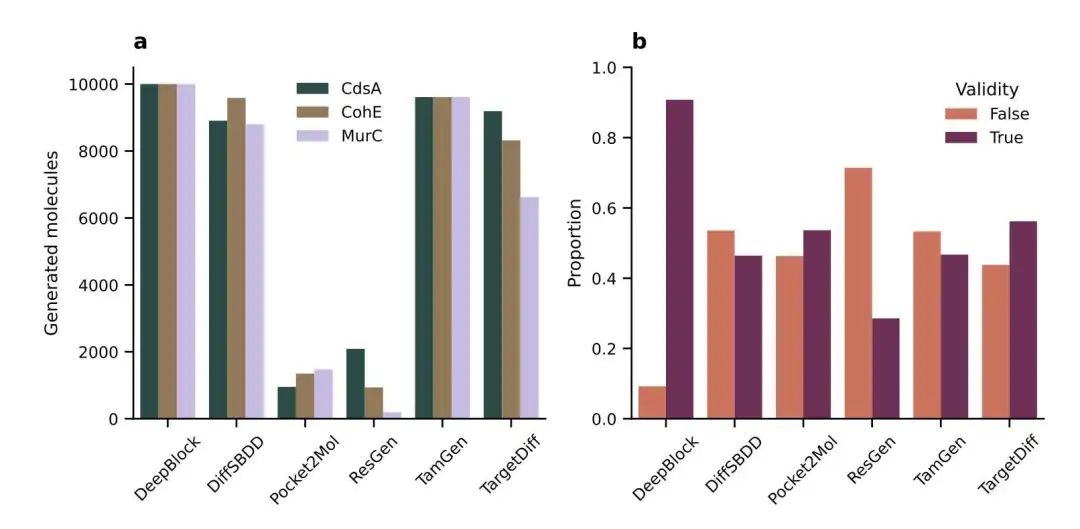
研究比较了六种结构感知生成模型:DeepBlock、DiffSBDD、Pocket2Mol、ResGen、TamGen 和 TargetDiff,评估指标包括安装、可用性、文档、输出格式和输入灵活性。DeepBlock 和 DiffSBDD 在易用性和实现方面得分最高,而 TamGen 和 TargetDiff 需要复杂且文档不足的预处理。DeepBlock 缺乏口袋特异性生成,但提供了用户友好的输出和内在结合位点检测。
六种模型共生成了超过 117,000 个分子。DeepBlock 实现了最高的化学有效性(约 90%),而其他模型的化学有效性约为 50%。ResGen 生成的有效分子最少(约 30%)。TamGen 探索了最广泛的化学空间,但也表现出较高的骨架冗余,经常在不同靶点上生成相同的分子,这表明其具有较强的先验性,但结构特异性较低。<
